Genetische Variation bei hawaiianischen Drosophila. IV. Allozyme Ähnlichkeit zwischen D. silvestris und D. heteroneura von der Insel Hawaii.
Maschinenübersetzung
Der Originalartikel ist in EN Sprache (Link zum Lesen) geschrieben.
Zusammenfassung
Die Arten sind endemisch auf der neuesten Insel des Archipels und sind weitgehend sympatrisch. Sie lassen sich morphologisch bei beiden Geschlechtern leicht unterscheiden. Mit standardisierten elektrophoretischen Verfahren haben wir 25 Loci untersucht, die für Strukturproteine von 539 silvestris und 325 heteroneura kodieren, die an drei weit auseinanderliegenden Standorten gesammelt wurden, an denen die beiden Arten sympatrisch sind. Paarweise Vergleiche innerhalb und zwischen den Arten zeigen die folgenden Koeffizienten der genetischen Identität (Neis I): innerhalb von silvestris 0.961 ± 0.01; innerhalb von heteroneura 0.949 ± 0.02; zwischen silvestris und heteroneura, 0.939 ± 0.01. Weder die Unterschiede innerhalb noch zwischen den Arten sind signifikant. Es gibt keine fixen allelischen Unterschiede innerhalb oder zwischen den Arten. An den drei Gebieten der Sympatrie zeigen die Arten Unterschiede in der Genfrequenz P < 0.05) an 9, 11 und 13 Loci respektive. Dies unterscheidet sich nicht wesentlich von der Variation innerhalb einer der Arten über die drei Standorte hinweg. Die beiden Arten haben ähnliche Heterozygotie (H) niveaus (silvestris, 0.083; heteroneura, 0.089) und Prozentsätze polymorpher Loci (beide 0.37). Es wird vorgeschlagen, dass trotz ihrer morphologischen Divergenz diese Arten viel neu gebildet sind als klassische Geschwisterarten. Signifikante allozymische Unterschiede haben möglicherweise nicht genügend Zeit gehabt, um sich anzusammeln.
DROSOPHILA silvestris und D. heteroneura sind ein Paar eng verwandter Riesenarten, die ausschließlich in den Regenwäldern der Insel Hawaii endemisch sind, der neuesten im Archipel. Sie sind in den meisten Gebieten sympatrisch, außer in höheren Lagen, wo die Populationen tendenziell ausschließlich silvestris sind.
Die Arten lassen sich morphologisch leicht unterscheiden (Hardy 1965), nicht nur durch den außergewöhnlich breiten Kopf beider Geschlechter von heteroneura, sondern auch durch die Farbmuster des Gesichts, des Thorax, der Pleuren und des Abdomens sowie durch Unterschiede im Muster der Flügelmarkierungen. D. silvestris trägt sechs einzigartige Inversionen im polymorphen Zustand; eine siebte Inversion ist polymorph innerhalb beider Arten (Carson und Stalker 1968; Craddock und Carson 1975). Es gibt keine festen Inversionen zwischen den Arten, und die beiden teilen sich einen gemeinsamen Satz von Standardsequenzen, d.h. sie sind chromosomal homosequentiell.
Obwohl es eine starke sexuelle Isolation zwischen den Arten in Labor-Kreuzungen gibt (Ahearn et al. 1974; Kaneshiro 1976), wurden F1-Hybriden aus beiden reziproken Kreuzungen erhalten (Craddok 1974; Ahearn und Val 1975). Beide Geschlechter dieser F1-Hybriden sind voll fruchtbar, und die genetische Segregation der morphologischen Artunterschiede wurde in der F1-Generation und in Rückkreuzungen beobachtet (Val 1976).
Vorläufige Studien zu Allozyten haben eine außergewöhnliche Ähnlichkeit zwischen den beiden Arten (S = 0,96; Johnson et al. 1975; Johnson und Carson 1975) gezeigt. Das vorliegende Papier erweitert die Untersuchung der genetischen Variabilität innerhalb und zwischen den beiden Arten auf lösliche Proteine, die von 25 Loci kodiert werden. Die Daten stammen aus drei Gebieten der Sympatrie. Die Ergebnisse bestätigen die auffällige biochemische Ähnlichkeit der Arten trotz der genetischen Unterschiede zwischen ihnen in Morphologie, Inversionen und Verhalten.
Materialien und Methoden
Im Jahr 1974 und 1975 sammelten wir die beiden Arten aus drei Gebieten mit moderater Höhe auf Hawaii (Die Große Insel), wo sie sympatrisch sind. Diese sind (Abbildung 1): (1) Kahuku Ranch im südlichen Teil des Kau-Distrikts (Höhe 1158 m); (2) Olaa, Wald in der Nähe der Vulkan-Experimentstation an der Wright Road, Puna-Distrikt (Höhe 1238 m) und (3) Pauahi, South Kona-Distrikt (Höhe 1311 m). Neu gefangene Männchen wurden so schnell wie möglich nach der Fang für die Elektrophorese verwendet. Weibchen wurden isoliert und durften F1-Nachkommen zur Verwendung in Speicheldrüsenpräparaten produzieren. Nach der Produktion von Larven wurden die Mütter für die Elektrophorese verwendet. Nur der Abdomen der Fliegen wurde der Elektrophorese unterzogen; der Rest (Kopf, Thorax und Anhänge) wurde montiert und in einer Sammlung platziert, die für morphologische Analysen verwendet werden sollte.
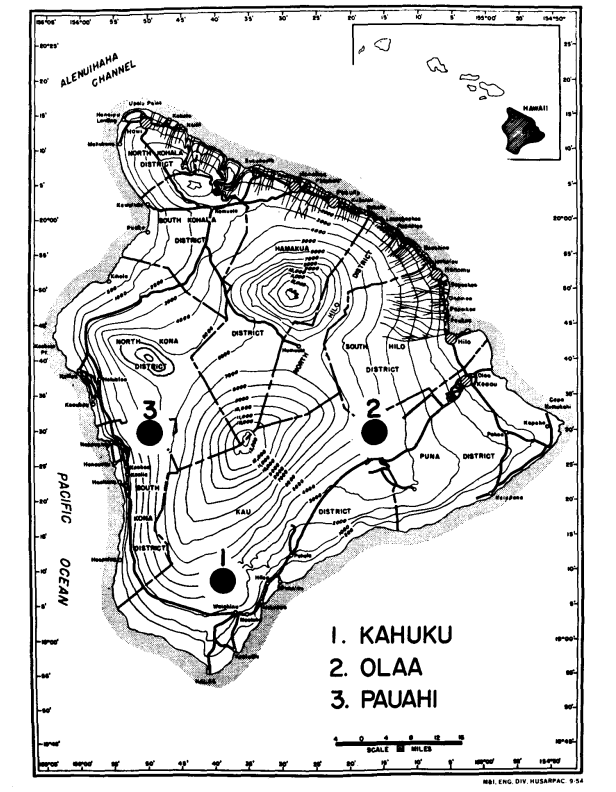
Nach der Entfernung wurde der Bauch über Nacht eingefroren und dann einzeln in 0,05 ml deionisiertem destilliertem Wasser homogenisiert, und jeder Überstand wurde von vier Wicks des Whatman Nr. I Filterpapiers (1 X 0,2 cm) absorbiert. Diese Wicks wurden dann getrennt und auf vier horizontale Stärkegele aufgetragen, die verschiedene Puffersysteme kombinierten. Nach der Elektrophorese wurde jedes Gel horizontal vier oder fünf Mal geschnitten, und diese Schnitte wurden separat für ein oder mehrere von 25 Enzymen gefärbt. Alle Arbeiten wurden mit Electrostarch Charge 303 bei 12% Konzentration durchgeführt.
Die drei in der Studie verwendeten Pufferkombinationen sind wie folgt: Puffersystem A = Gel-Puffer: pH 8.9, 0.0076 m Tris, 0.005 m Citrate; Elektrodenpuffer: pH 8.7, 0.269 m Borat, 0.1 m Natriumhydroxid. Puffersystem C = Gel-Puffer: pH 8.1, 0.074 m Tris, 0.008 m Citrate; Elektrodenpuffer: pH 8.1, Kathode = 0.343 m Tris, 0.079 m Citrate; Anode = 0.458 m Tris, 0.0104 m Citrate. Puffersystem JRP = Gel-Puffer: pH 7.0, 0.009 m Tris, 0.003 m Citrate; Elektrodenpuffer: 0.135 m Tris, 0.040 m Citrate. Alle diese Puffer und die verwendeten Färbemethoden sind ähnlich wie die von Steiner und Johnson (1973) sowie Ayala et al. (1972; 1974a) beschriebenen.
Die Allele an einem bestimmten Locus wurden entsprechend den relativen Mobilitäten ihrer Allozyme in den angegebenen elektrophoretischen Puffersystemen nummeriert. Das Allel, das das häufigste Allozyme erzeugte, wurde mit l.00 bezeichnet, und Allele, die schnellere oder langsamer migrierende Banden entsprachen, wurden willkürlich Werte zugewiesen, die ihre jeweiligen Mobilitäten anzeigten.
Das verwendete Puffer-System und die Notation für jedes Enzym sind wie folgt: Puffer-System A : Esterase (EST, 1 Locus) ; Alkalische Phosphatase (AP, 1 Locus) ; Saure Phosphatase (ACPH, 1 Locus) ; Glutamat-Oxalacetat-Transaminase (GOT, 2 Loci) ; Aldehydoxidase (AO, 1 Locus) ; Octanol-Dehydrogenase (ODH, I Locus) ; Alkohol-Dehydrogenase (ADH, 1 Locus) ; Xanthin-Dehydrogenase (XDH, 1 Locus) ; Leucin-Aminopeptidase (LAP, 2 Loci) ; Glycerinaldehyd-3-Phosphat-Dehydrogenase (G-3-PDH, 1 Locus). Puffer-System C : Malat-Enzym (ME, 1 Locus) ; Malat-Dehydrogenase (MDH, 2 Loci) ; α-Glycerophosphat-Dehydrogenase (α-GPDH, 1 Locus) ; Phosphoglucomutase (PGM, I Locus) ; Fumarase (FUM, 1 Locus) ; Sorbitol-Dehydrogenase (SDH, 1 Locus) ; Hexokinase (HK, 2 Loci) ; Isocitrat-Dehydrogenase (IDH, 1 Locus). Puffer-System JRP : 6-Phosphogluconat-Dehydrogenase (6-PGDH, 1 Locus) ; Hydroxybutyrat-Dehydrogenase (HBDH, 1 Locus) ; Phosphoglucose-Isomerase (PGI, 1 Locus).
Ergebnisse
Die grundlegenden Alloenzymdaten sind in Tabelle 1 angegeben. Tabelle 2 zeigt die Ergebnisse der Verwendung der Daten in Tabelle 1 zur Berechnung der Koeffizienten der genetischen Ähnlichkeit, I, und Distanz, D (Nei 1972) in paarweisen Vergleichen innerhalb und zwischen den Arten. In derselben Tabelle (unterhalb der Diagonalen) sind die Anzahl der Loci dargestellt, die Unterschiede in der Genfrequenz (P ≤ 0,05) in paarweisen Vergleichen aufweisen. Die Berechnung wurde unter Verwendung von χ2 durchgeführt, indem die Frequenz des häufigeren Allels gegen eine gepoolte Frequenz der anderen Allele verwendet wurde. Die Korrektur nach Yates wurde angewendet.
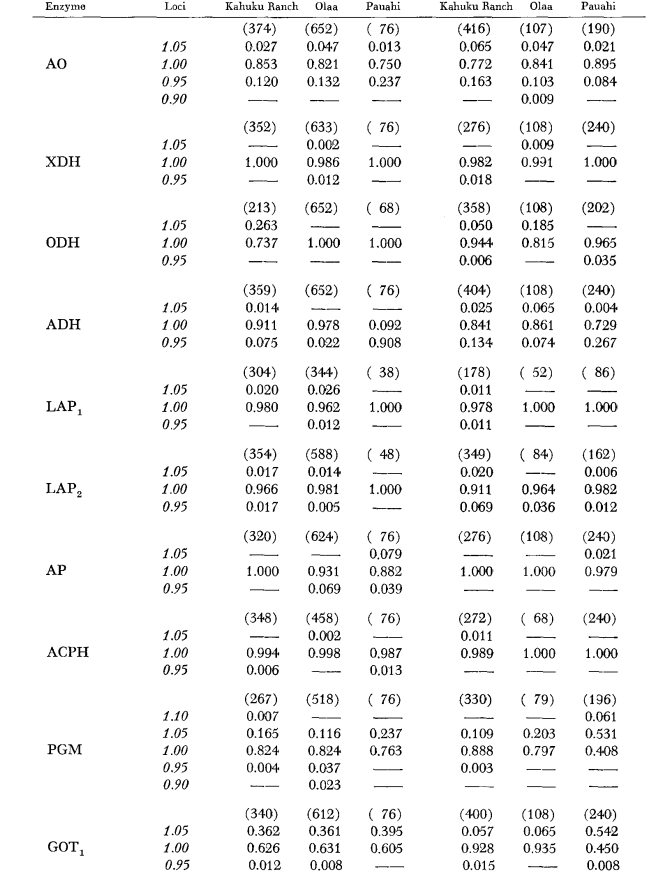
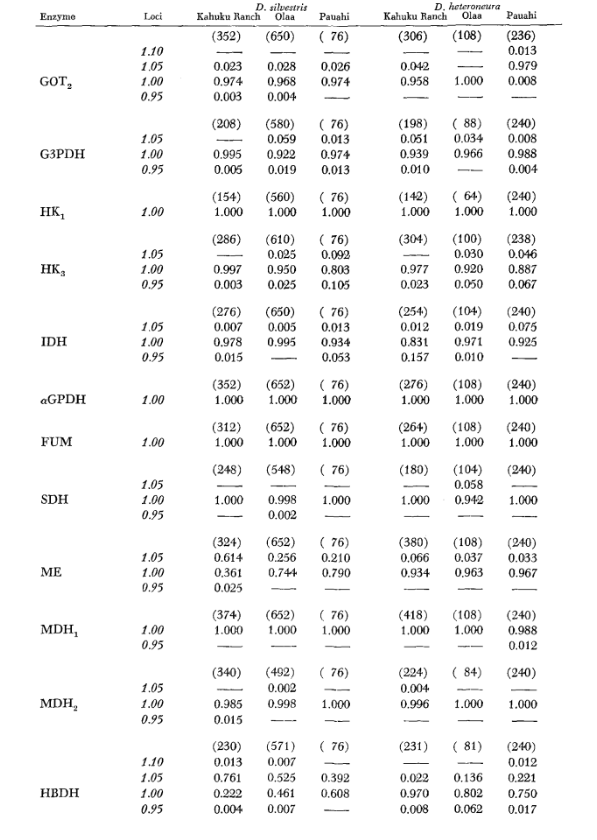
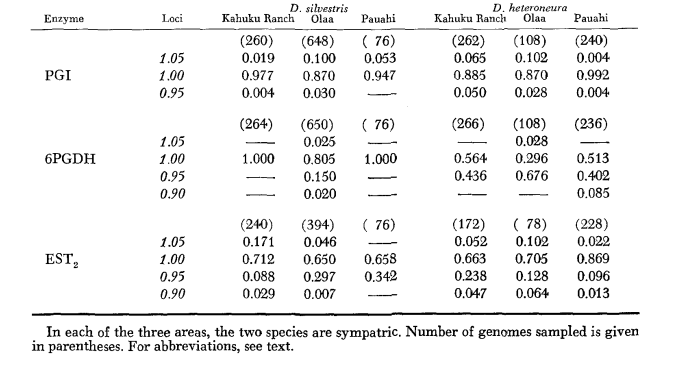
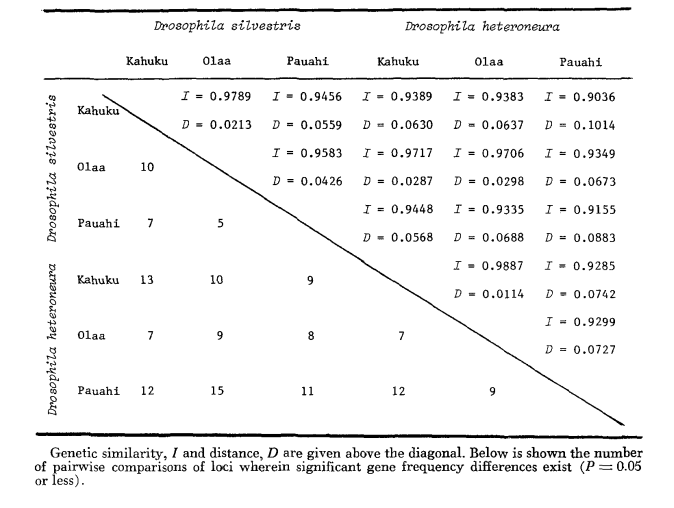
Durchschnittliche Heterozygotie-Niveaus und Schätzungen der Häufigkeit polymorpher Loci sind in Tabelle 3 angegeben. Eine grafische Darstellung der Häufigkeit des Allels 1.0 für jedes Alloenzym in jeder Population ist in Abbildung 2 dargestellt.
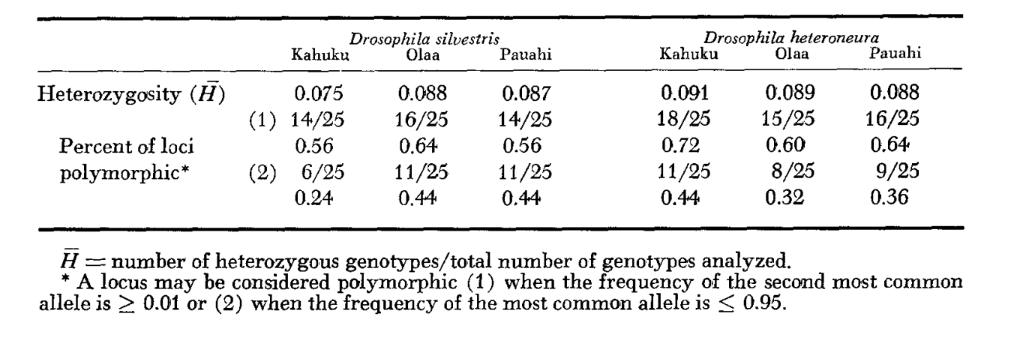
Die allozymische Ähnlichkeit ist sowohl innerhalb als auch zwischen den Arten sehr groß, unabhängig davon, ob sie durch Ähnlichkeitskoeffizienten oder durch Unterschiede in der Genfrequenz gemessen wird (siehe Zusammenfassung in Tabelle 4). Es gibt keine festen allelischen Unterschiede, weder innerhalb noch zwischen den Arten. An den drei Gebieten der Sympatrie zeigen die Arten Unterschiede in der Genfrequenz (P = 0.05) an 9, 11 und 13 Loci. Dies unterscheidet sich nicht wesentlich von der Variation innerhalb einer der Arten über die drei Lokalitäten (silvestris: 5, 7, 10; heteroneura: 7, 9, 12). Die beiden Arten haben ähnliche Heterozygotie-Niveaus (silvestris 0.083; heteroneura 0.089) und Prozentsätze der polymorphen Loci (beide 0.37).

Einige Details sind von Interesse. Die Ähnlichkeit zwischen den Arten ist am geringsten in Pauahi (Tabelle 2; Abbildung 2). Die Inspektion zeigt, dass dies hauptsächlich auf die Häufigkeitsunterschiede bei ADH und GOT2 zurückzuführen ist. Große Unterschiede an diesen Loci erscheinen zwischen den Arten nur in diesem Gebiet; das heißt, das 1.00 GOT2-Allele ist nur in Pauahi heteroneura niedrig und das 1.00 -Allele von ADH ist nur in Pauahi silvestris niedrig (Tabelle 1; Abbildung 2).
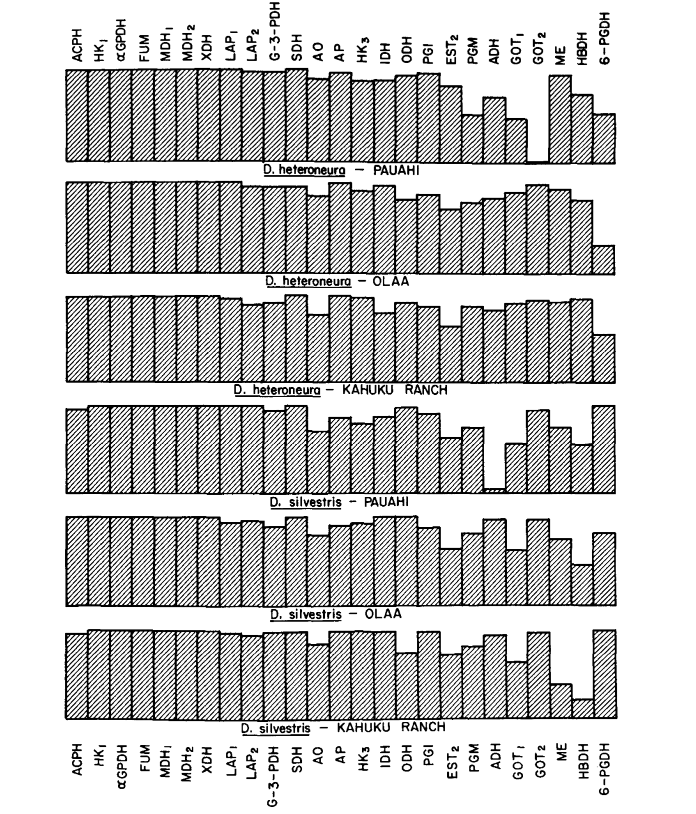
Die Ähnlichkeit zwischen den Arten ist im Kahuku-Gebiet etwas größer. Hier betreffen die Unterschiede hauptsächlich GOT1, ME und HBDH. In Olaa ist die Ähnlichkeit zwischen den Arten am größten. GOT1 und HBDH zeigen erneut Unterschiede, obwohl diese nicht ganz so groß sind wie die zwischen den Arten in Kahuku.
Diskussion
Genetische Vergleiche von Populationen unter Verwendung von Alloenzymen haben in den letzten zehn Jahren die Literatur überschwemmt (siehe die Übersichten von Ayala 1975 und Powell 1975). Da sowohl die untersuchten Proteine als auch die verwendeten elektrophoretischen Methoden im Großen und Ganzen ähnlich sind, können Ergebnisse aus recht unterschiedlichen Taxa sinnvoll verglichen werden. Zum Beispiel kann die genetische Ähnlichkeit zwischen zwei Arten von Eidechsen, wenn sie auf einen Koeffizienten wie den von Nei reduziert wird, direkt mit ähnlichen Daten von Artenpaaren von Mäusen, Pflanzen oder Drosophila verglichen werden.
In ihrem wegweisenden Artikel wiesen Lewontin und Hubby (1966) auf eine Reihe von Gründen hin, warum die standardmäßigen elektrophoretischen Methoden dazu neigen, die genetische Variabilität und den genetischen Unterschied zu unterschätzen. Dies muss heute umso mehr berücksichtigt werden, da sich die sequenzielle elektrophoretische Analyse kürzlich entwickelt hat. So haben Singh, Lewontin und Felton (1976) sowie Coyne (1976) gezeigt, dass die Anzahl der Allele am Locus der Xanthin-Dehydrogenase von D. pseudo-obscura und persimilis in recht kleinen natürlichen Proben dieser Arten mindestens viermal größer ist als die Anzahl, die durch standardmäßige Methoden offenbart wird. Was interpopulationale Vergleiche betrifft, scheinen diese neuen Methoden sicher die beobachteten Ähnlichkeiten auf allen Ebenen der Populationsdifferenzierung (z. B. lokale Populationen, Unterarten, Semispezies, Arten) zu reduzieren.
Trotz dieser neuen Entwicklungen sind Vergleiche, die auf den Standardmethoden basieren, weiterhin wertvoll, wenn ihre Einschränkungen erkannt werden. D. silvestris und heteroneura sind allozymisch viel näher beieinander als jede andere Drosophila-Artenpaar, das bisher untersucht wurde, einschließlich aller Paare von „Geschwister“-Arten, Unterarten und Semispezies (Ayala et al. 1974b). Tatsächlich sind die Ähnlichkeitskoeffizienten für Populationen innerhalb von silvestris oder heteroneura von der gleichen Größenordnung wie die zwischen denselben beiden Arten. Die kleinen Unterschiede, die zwischen Populationen der beiden Arten existieren, sind darüber hinaus nicht in den drei untersuchten Gebieten gleich; das heißt, sie betreffen nicht dieselben Loci oder Allele.
Wie in der Einleitung besprochen, erkennen wir diese beiden Entitäten weiterhin als gute biologische Arten an. Sie sind morphologisch unterschiedlich, weit verbreitet sympatrisch, verhaltensmäßig isoliert und unterscheiden sich in ihren chromosomalen Polymorphismen. Die morphologischen Unterschiede hängen von ungefähr vierzehn Polygenen ab (Val 1976).
Ein auffälliges und wichtiges Merkmal dieser Arten ist jedoch die Tatsache, dass reziproke Hybriden, die im Labor gewonnen wurden, in beiden Geschlechtern vollständig fruchtbar sind (Craddock 1974; Ahearn und Val 1975). Dementsprechend muss die natürliche Hybridisierung in Betracht gezogen werden. Obwohl Craddock (1974) dafür keine Beweise fand, berichteten Kaneshiro und Val (1977), dass etwa zwei Prozent der in Kahuku Ranch gefangenen Wildfliegen morphologisch als Hybriden identifiziert werden können. Dies ist dasselbe Gebiet, in dem natürliche Hybriden zwischen zwei anderen Arten gefunden wurden (Carson, Nair und Sene 1975). Die morphometrische Analyse der Kopfform wurde erfolgreich eingesetzt, um die genaue reziproke Abstammung bestimmter F1-Hybriden zu bestimmen. In einem Fall produzierte ein wild gefangenes D. silvestris-Weibchen Nachkommen, die ausschließlich aus F1-Hybriden bestanden. Da jedes wilde Weibchen sofort nach der Fang von allen Männchen isoliert wurde, ist klar, dass die relevanten Paarungen in der Natur stattfanden. Es sollte betont werden, dass der Hintergrund umfangreicher morphometrischer Studien zu Laborhybriden (Val 1976) die Grundlage für diese neueren natürlichen Beobachtungen geschaffen hat und daher die Diagnose dieser Fälle auf soliden vergleichenden Beweisen beruht. Alle natürlichen Hybridindividuen wurden aus den in diesem Papier präsentierten elektrophoretischen Daten ausgeschlossen.
Ein weiterer Fakt, der zur ungewöhnlichen Situation dieser Arten beiträgt, ist ihre enge ökologische Ähnlichkeit. Beide wurden aus denselben einzelnen verrottenden Stängeln der gemeinsamen Wirtspflanze, Clermontia, gezüchtet. Beide bilden ebenfalls ihre Leks an sehr ähnlichen Orten, sodass sich Männchen und Weibchen beider Arten in der Natur interagieren.
Was ist der Grund für die sehr große allozymische Ähnlichkeit dieser Arten? Zwei Möglichkeiten bieten sich an. Erstens könnte es sein, dass die Divergenz zwischen den Arten ursprünglich mit einem gewissen Maß an elektroforetischer Divergenz einherging. Die derzeit beobachteten Ähnlichkeiten würden somit erfordern, dass solche Unterschiede durch introgressive Hybridisierung nach der Artbildung erodiert wurden. Eine zweite Ansicht besagt, dass nur geringfügige elektroforetische Veränderungen den Artbildungsereignissen ursprünglich beigefügt waren. Dies könnte mit der sehr erheblichen ökologischen Nähe korreliert sein.
Wir favorisieren die zweite Ansicht aus mehreren Gründen. Die beiden Arten sind am Olaa am ähnlichsten. Dennoch wurden starke Beweise für eine kürzliche Hybridisierung nur für Kahuku erhalten, wo die Arten allozymisch am weitesten entfernt sind. In Kahuku zeigen die natürlichen Populationen außerdem keine Hinweise darauf, dass sie einen „Hybrid-Schwarm“ bilden, der der breiten genetischen Rekombination ähnelt, die in Labor-Kreuzungen beobachtet wird. Tatsächlich findet wahrscheinlich eine Introgression zwischen den Arten statt, aber sie könnte durch natürliche Selektion auf ein enges genotypisches Spektrum beschränkt sein. Somit könnten nur Gene von biologisch geringer Bedeutung (das „offene System“, Carson 1975) in der Lage sein, über die Artgrenze zu fließen. Umgekehrt könnten andere Gene ein Merkmal oder einen funktionalen Teil eines geschlossenen Systems epistatischer Gleichgewichte darstellen, das einzigartig für eine oder andere der Arten ist. Selektion könnte somit dazu dienen, die Aufspaltung eines solchen „geschlossenen Systems“ (Carson 1975) zu verhindern.
Aufgrund der geologischen Neuheit der Insel Hawaii sind wir geneigt zu vermuten, dass D, silvestris und D. heteroneura tatsächlich neu gebildet sein könnten. Diese Umstände könnten die primäre Ursache für die elektrophoretische Ähnlichkeit der beiden Arten sein. In der Tat könnten sie viel neuer sein als klassische „Geschwister“-Artenpaare (siehe Carson 1976b). Wie bei anderen solchen scheinbaren Neospezies, (z.B. Gottlieb 1974; Turner 1974; Avise, Smith und Ayala 1975), war die allozymische Differenzierung relativ gering. In der Tat besteht die Möglichkeit, dass die allozymische Differenz einfach mit der seit dem kladistischen Ereignis vergangenen Zeit korreliert, die die beiden verglichenen Entitäten getrennt hat (Avise, Smith und Ayala 1975; Carson 1976a). Morphologische Unterschiede hingegen könnten als bessere Indikatoren für die zugrunde liegenden Effekte von regulatorischen Genen dienen, die die wichtigen genetischen Unterschiede zwischen neu gebildeten Arten kennzeichnen.
Autoren: F. M. Sene, H. L. Carson
Zitierte Literatur:
- Ahearn, J. N., H. L. Carson, Th. Dobzhansky und K. Y. Kaneshiro, 1974 Ethologische Isolation zwischen drei Arten der planitibia-Untergruppe von Hawaiian Drosophila. Proc. Natl. Acad. Sci. U.S. 71: 901—903.
- Ahearn, J. N. und F. C. Val, 1975 Fruchtbare interspezifische Hybriden von zwei sympatrischen Hawaiian Drosophila. Genetics 80: s9.
- Avise, J. C., J. J. Smith und F. J. Ayala, 1975 Adaptive Differenzierung mit wenig genischem Wandel zwischen zwei einheimischen Kalifornischen Minows. Evolution '29: 411—426.
- AYALA, F. J., 1975 Genetische Differenzierung während des Speziationsprozesses. Evolutionäre Biologie 8: 1—78.
- Ayala, F. J., J. R. Powell, M. L. Tracey, C. A. Mourão und S. Pérez-Salas, 1972 Enzymvariabilität in der Drosophila willistoni-Gruppe. IY Genetische Variation in natürlichen Populationen von Drosophila willistoni. Genetics 70: 113—139.
- Ayala, F. J., M. L. Tracey, L. G. Barr und J. G. Ehrenfeld, l9f4a Genetische und reproduktive Differenzierung der Unterart, Drosophila equinoxialis caribbensis. Evolution 28: 24—41.
- Ayala F. J., M. L. Tracey, D. Hedgecock und R. C. Richmond, 1974b Genetische Differenzierung während des Speziationsprozesses in Drosophila. Evolution 28: 576—592.
- Carson, H. L. 1975 Die Genetik der Speziation auf diploidem Niveau. Am. Naturalist 109: 83—92, 1976a Schlussfolgerung über die Entstehungszeit einiger Drosophila-Arten. Nature 259: 395—396, 1976b Genetische Unterschiede zwischen neu gebildeten Arten. Bioscience 26: 700—701.
- Carson, H. L., P. S. Nair und F. M. Sene, 1975 Drosophila-Hybriden in der Natur: Beweis für den Genaustausch zwischen sympatrischen Arten. Science 189: 806—807.
- Carson, H. L. und H. D. Stalker, 1968 Polytene Chromosomenbeziehungen in Hawaiianischen Arten von Drosophila. II. Die D. planitibia-Untergruppe. Univ. Texas Publ. 6818: 355-365.
- Coyne, J. A., 1976 Mangel an genetischer Ähnlichkeit zwischen zwei Geschwisterarten von Drosophila, wie durch verschiedene Techniken gezeigt. Genetics 84: 593—607.
- Craddock, E. M., 1974 Reproduktive Beziehungen zwischen homosequenten Arten von Hawaiian Drosophila. Evolution 28: 593—606.
- Craddock, E. M. und H. L. Carson, 1975 Chromosomenvariabilität in einer endemischen Hawaiian Drosophila-Art. Genetics 80: s23.
- Gottlieb, L. D., 1974 Genetische Bestätigung des Ursprungs von Clarkia lingulata. Evolution 2B: 244-250.
- Hardy, D. E., 1965 Insekten von Hawaii, Bd. 12. S. 814. University of Hawaii Press, Honolulu.
- Johnson, W. E. und H. L. Carson, 19f5 Allozyme-Variation in Drosophila silvestris. Genetics 80: s46.
- Johnson, W. E., H. L. Carson, K. Y. Kaneshiro, W. W. M. Steiner und M. M. Cooper, 1975 Genetische Variation in Hawaiian Drosophila. II. Allozyme-Differenzierung in der D. planitibia-Untergruppe. S. 563—584. In: Isozyme IV. Genetik und Evolution. Herausgegeben von C. L. Markert. Academic Press, New York.
- Kaneshiro, K. Y., 1976 Ethologische Isolation und Phylogenie in der planitibia-Untergruppe von Hawaiian Drosophila. Evolution 30: 740-745.
- Kaneshiro, K. Y. und F. C. Val, l977 Natürliche Hybridisierung zwischen einem sympatrischen Paar von Hawaiian Drosophila. Am. Naturalist (in Druck).
- Lewontin, R. C. und J. L. Hubby, 1966 Ein molekularer Ansatz zur Untersuchung der genetischen Heterozygotie in natürlichen Populationen. II. Menge der Variation und Grad der Heterozygotie in natürlichen Populationen von Drosophila pseudoobscura. Genetics 54: 595—609.
- Nei, M., 1972 Genetische Distanz zwischen Populationen. Am. Naturalist 106: 283—292.
- Powell, I. R., 1975 Proteinvariabilität in natürlichen Populationen von Tieren. Evol. Biol. 8: 79-113.
- Simon, R. S., R. C. LEWONTIN und A. A. Fzczou, 19 Z6 Genetische Heterogenität innerhalb elektro- phoretischer “Allele” von Xanthin-Dehydrogenase in Drosophila pseudoobscura. Genetics 84: 609—629.
- Steiner, W. W. M. und W. E. Johnson, 1973 Techniken für die Elektrophorese von Hawaiian Drosophila. US/IBP Island Ecosystems IRP. Tech. Report 30: ii + 21 S.
- Turner B. J., 1974 Genetische Divergenz von Death Valley-Pfahlfischarten: Biochemische versus morphologische Beweise. Evolution fl8: 281-294.
- Val, F. C., 1976 Genetik der morphologischen Unterschiede zwischen zwei interfertilen Arten von Drosophila. Genetics 83: s78.