Geringe Satelliten-DNA-Variabilität in natürlichen Populationen von Drosophila antonietae, die an verschiedenen evolutionären Ereignissen beteiligt sind
Maschinenübersetzung
Der Originalartikel ist in EN Sprache (Link zum Lesen) geschrieben.
Drosophila antonietae ist eine cactophile Art, die im mesophilen Wald des Paraná–Paraguay-Flussbeckens und in den Dünen der Südatlantikküste Brasiliens vorkommt. Obwohl die genetische Struktur der Populationen im Paraná–Paraguay-Flussbecken bereits etabliert wurde, ist die Beziehung zwischen diesen Populationen und denen an der Atlantikküste umstritten. In dieser Studie verglichen wir 33 repetitive Einheiten der pBuM-2 Satelliten-DNA, die von Individuen aus 8 Populationen von D. antonietae in diesen geografischen Regionen isoliert wurden, einschließlich einiger Populationen, die sich innerhalb einer Kontaktzone mit dem eng verwandten D. serido befinden. Die pBuM-2 Sequenzen zeigten eine geringe interpopulationale Variabilität. Dieses Ergebnis wurde als Folge sowohl des Genflusses zwischen den Populationen als auch des ungleichen Crossing-Over interpretiert, das die Homogenisierung der Tandemarrays fördert. Die hier präsentierten Ergebnisse, zusammen mit denen früherer Studien, heben die Verwendung von pBuM-2 zur Lösung taxonomischer Konflikte innerhalb des D. buzzatii Artenclusters hervor.
Einführung
Drosophila antonietae ist ein Mitglied des D. buzzatii Artenclusters, einer monophyletischen Gruppe, die aus 7 Geschwisterarten besteht, die natürlich endemisch in der neotropischen Region vorkommen und in den offenen und trockenen Wäldern im östlichen Südamerika zu finden sind (Manfrin und Sene 2006). Diese Arten nutzen nekrotisches Kaktusgewebe als Brutstätten (Pereira et al. 1983); deshalb ist ihr geografisches Verbreitungsgebiet auf den Verbreitungsbereich der Wirtkaktusse beschränkt. Somit ist D. antonietae ein attraktives Modell, um evolutionäre Prozesse zu studieren (Manfrin und Sene 2006).
Die geografische Verbreitung von D. antonietae kann auf 2 Arten charakterisiert werden. In den Tälern, die vom Paraná–Paraguay-Flussbecken umschlossen sind, finden sich Populationen in mesophilen Wäldern, die mit dem Kaktus Cereus hildemannianus assoziiert sind (Tidon-Sklorz und Sene 2001; Mateus und Sene 2003). In seiner südöstlichen Verbreitung kommt D. antonietae in den Dünenregionen der Atlantikküste vor, wobei hauptsächlich C. hildemannianus, aber auch Kaktusse der Gattung Opuntia genutzt werden (Ruiz et al. 2000; Manfrin und Sene 2006) (Abbildung 1).
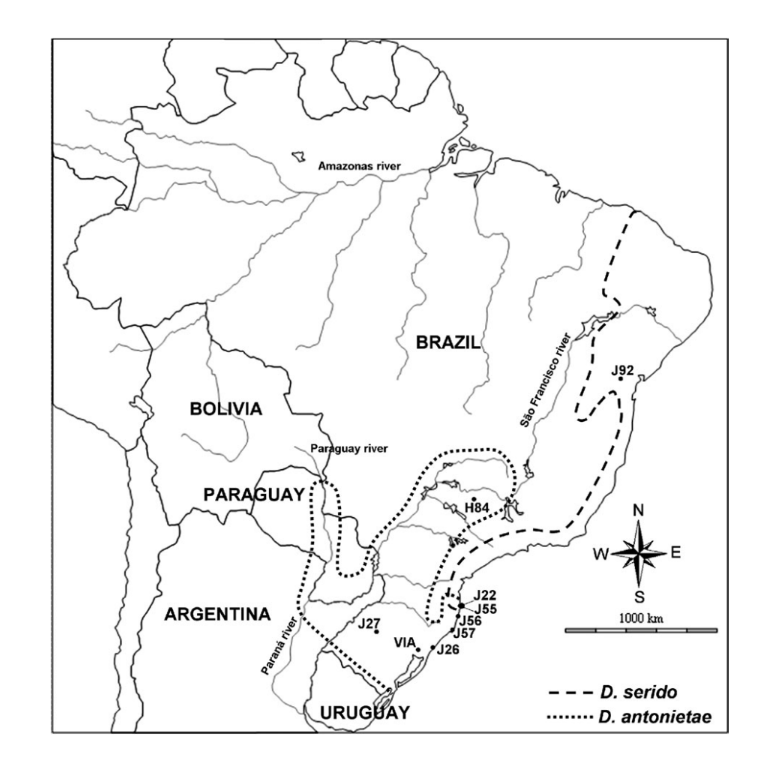
Frühere Studien haben vorgeschlagen, dass Populationen von D. antonietae aus dem Paraná–Paraguay-Flussbecken gemäß dem Modell der Isolation durch Distanz strukturiert sind, aufgrund der positiven Korrelation zwischen morphologischen und genetischen Distanzen mit geografischen und ökologischen Distanzen (letztere definiert als die Distanz zwischen 2 Populationen entlang des Flussverlaufs) (de Brito et al. 2002; Mateus und Sene 2007). Basierend auf der Anzahl der Migranten pro Generation schlugen Mateus und Sene (2007) einen moderaten Einfluss sowohl des Genflusses als auch der genetischen Drift in diesen Populationen von D. antonietae vor, wobei der Genfluss die genetische Drift überlappt und einen genetischen Gradient fördert.
Eine phylogeografische Analyse basierend auf mitochondrialer DNA deutete darauf hin, dass die Verbreitung von D. antonietae im Südosten, an der Atlantikküste Brasiliens, durch Expansionsereignisse von D. antonietae-Populationen aus der zentralen Depression des Bundesstaates Rio Grande do Sul kolonisiert wurde (de Brito et al. 2002; Manfrin und Sene 2006). Diese Analyse konnte jedoch nicht bestimmen, ob dieser Expansion eine Aufrechterhaltung des Genflusses folgte, mit der Erwerbung einer Populationsstruktur gemäß dem Modell der Isolation durch Distanz, oder ob sie von Fragmentierung gefolgt wurde, was auf das Fehlen von Genfluss zwischen den Populationen der Atlantikküste und denen aus dem Paraná–Paraguay-Flussbecken hindeutet (de Brito et al. 2002). Die Daten zur Chromosomeninversion könnten auf einen gewissen Grad an Fragmentierung zwischen den Populationen aus diesen 2 unterschiedlichen Umgebungen hindeuten, da die Populationen aus den Tälern des Paraná–Paraguay-Flussbeckens polymorph für die Inversionen 2y8, 2z8 und 5e sind, während die Populationen von der Atlantikküste polymorph für 2z8 sind (Ruiz et al. 2000). Die Atlantikküstenpopulationen von D. antonietae haben eine weitere wichtige Eigenschaft: Sie stehen in Kontakt mit der eng verwandten Art D. serido (D. buzzatii-Cluster) (Ruiz et al. 2000; Manfrin und Sene 2006). Dieses Überlappungsgebiet ist das Ergebnis der Expansion von D. serido von der Nord- zur Südatlantikküste und teilt sich einen geografischen Standort mit
D. antonietae Populationen (de Brito et al. 2002; Manfrin und Sene 2006) (Abbildung 1). Morphologische Merkmale und Analysen der mitochondrialen DNA deuten darauf hin, dass eine Hybridisierung zwischen
D. antonietae und D. serido innerhalb der Kontaktzone stattgefunden hat (C.K.B. Santos und Kollegen, unveröffentlichte Daten).
Satelliten-DNA sind hochgradig repetitive, tandemartig angeordnete DNA-Sequenzen, die einen wesentlichen Bestandteil des Heterochromatins eukaryotischer Organismen darstellen (Charlesworth et al. 1tt4; Palomeque und Lorite 2008). Satelliten befinden sich typischerweise in der Nähe von zentromerischen Regionen und seltener in Telomeren (Ugarkovic und Plohl 2002; Kuhn et al. 2008). Diese Sequenzen entwickeln sich auf konzertierte Weise, während die Hauptmechanismen, die an ihrer Evolution beteiligt sind, Slippage-Replikation, ungleiche Crossing-over, Genkonversion und Rolling-Circle-Replikation sind (Dover 1982; Charlesworth et al. 1994).
Einige der Satelliten-DNA-Familien entwickeln sich schnell, was ihre Verwendung als molekulare Marker in interspezifischen Vergleichsstudien ermöglicht (Watabe et al. 1997; Picariello et al. 2002; Kuhn und Sene 2005). Die Verwendung dieser Sequenzen als molekulare Marker in Populationsstudien wurde jedoch relativ wenig erforscht, obwohl berichtet wurde, dass eine konzertierte Evolution in Populationen von Cyprinodon variegates (Cyprinodontidae) (Elder und Turner 1994) und Acrossocheilus paradoxus (Cyprinidae) (Wu et al. 1999) stattgefunden hat.
Drei Satelliten-DNA-Familien, die unter den Arten des D. buzzatii-Clusters geteilt werden, wurden formal beschrieben: pBuM (Kuhn et al. 1999, 2008; Kuhn und Sene 2005), DBC-150 (Kuhn et al. 2007) und SSS139 (Franco et al. 2008). Die pBuM-Familie besteht aus 2 Unterfamilien, pBuM-1 und pBuM-2 (Kuhn und Sene 2005; Kuhn et al. 2008). Die Unterfamilie pBuM-2 besteht aus AT-reichen Monomeren von 370 bp, die qualitative (feste Nukleotidsubstitution) und quantitative (Kopienzahl) Unterschiede zwischen den Arten des D. buzzatii-Clusters aufweisen (Kuhn und Sene 2005; Kuhn et al. 2007, 2008).
In dieser Arbeit demonstrieren wir die intra-spezifische Variabilität der pBuM-2 Satelliten-DNA in D. antonietae, unter Verwendung von Populationen aus 2 unterschiedlichen Umgebungen (dem Paraná–Paraguay-Becken und der Atlantikküste), die unterschiedliche evolutionäre Geschichten aufweisen, die mit der Aufrechterhaltung des Genflusses im ersteren und Hybridisierungsereignissen mit der eng verwandten Art D. serido im letzteren verbunden sind. Wir haben uns entschieden, die pBuM Satelliten-DNA als molekulares Marker zu verwenden, da sie 14 diagnostische Nukleotid-Substitutionen zwischen D. antonietae und D. serido aufweist (Kuhn und Sene 2005). Somit ermöglicht pBuM die taxonomische Identifizierung von Exemplaren, die aus der Kontaktzone isoliert wurden. Darüber hinaus sind die evolutionären Dynamiken von Satelliten-DNA-Sequenzen auf intra-spezifischer Ebene nicht vollständig verstanden, sodass weitere empirische Daten zu einem besseren Verständnis des Prozesses beitragen könnten, der der Evolution der Satelliten-DNA zugrunde liegt (Ugarkovic und Plohl 2002; Palomeque und Lorite 2008).
Material und Methoden
Proben
Wir haben 8 brasilianische Populationen von D. antonietae (Abbildung 1) untersucht. Drei dieser Populationen befinden sich in den Tälern des Paraná–Paraguay-Flussbeckens: J2/ (Santiago, Bundesstaat Rio Grande do Sul), Via (Viamão, Bundesstaat Rio Grande do Sul) und H84 (Serrana, Bundesstaat São Paulo, Zugangsnummer AY656609– AY656615; Kuhn und Sene 2005); und 5 befinden sich in den Dünen der Atlantikküste, innerhalb der Kontaktzone mit
D. serido: J56 (Garopaba, Bundesstaat Santa Catarina), J55 (Armaxcão Strand, Florianópolis, Bundesstaat Santa Catarina), J22 (Joaquina Strand, Florianópolis, Bundesstaat Santa Catarina), J5/ (Laguna, Bundesstaat Santa Catarina) und J26 (Osório, Bundesstaat Rio Grande do Sul) (Abbildung 1). Die Sequenzen von D. antonietae wurden zusammen mit G pBuM-2-Sequenzen von D. serido (Population Jf2—Milagres, Bundesstaat Bahia, Zugangsnummer AY656616–AY656621; Kuhn und Sene 2005) analysiert, um einige Introgressionsereignisse in Populationen von der Atlantikküste zu identifizieren.
Isolation von pBuM-2 Satelliten-DNA und Sequenzanalyse
Genomische DNA von einem männlichen Individuum (wild gefangene Fliegen) pro Population wurde mit dem Wizard Genomic DNA Purification Kit (Promega) extrahiert. Die genomische DNA wurde einer Polymerase-Kettenreaktion unterzogen, um die Kopien der pBuM-2 Familie [Primer A2F (CGGAGTA-TTTTTCATTCGAC) und A2R (GGTATGCCATAAAG-AAGTCG)] gemäß Kuhn et al. (2008) zu amplifizieren. Die resultierenden Banden von etwa 400 bp wurden durch Übernachtinkubation in einer Elutionslösung (500 mM NaAc; 1 mM Ethylendiamintetraessigsäure) aus dem Gel eluiert. Die zurückgewonnenen Fragmente wurden mit dem pMOSBlue blunt ended Klonierungs-Kit (RPN 5110; Amersham Pharmacia Biotech) kloniert. Plasmid-DNA wurde gemäß der in Sambrook et al. (1989) beschriebenen Methodik vorbereitet, und die DNA-Vorlagenreaktion für die Sequenzierung wurde gemäß dem Handbuch des BigDye Terminator Cycle Sequencing Ready Reaction Kit (PerkinElmer) vorbereitet. Die automatische DNA-Sequenzierung wurde auf einem ABI Prism 377 Sequencer (PerkinElmer) durchgeführt.
Sequenzalignierungen wurden in CLUSTALW 1.8 (Thompson et al. 1994) durchgeführt und in BioEdit (Hall 1999) bearbeitet. Wir verwendeten den Algorithmus und das statistische Modell, das von Betrán et al. (1997) vorgeschlagen wurde, um Gene-Konversionsstrecken zu erkennen. Der Parameter Rm, der die minimale Anzahl von Rekombinationsevents in einer DNA-Probe schätzt (Hudson und Kaplan 1985), wurde verwendet, um Rekombinationsevents zu erkennen. Obwohl dieser Parameter nicht speziell ungleiche Crossing-Over misst, ist es vernünftig anzunehmen, dass Rm Werte größer als null hätte, wenn ungleiche Crossing-Over stattgefunden hätte. In diesem Sinne verwenden wir Rm als indirekten Beweis für ungleiches Crossing-Over. Die genetischen Distanzen wurden gemäß p-Distanz berechnet, die den Anteil der Nukleotidunterschiede zwischen 2 Sequenzen darstellt (d.h. p5 Np/N, wobei Np die Unterschiede der Nukleotide und N die Gesamtzahl der Nukleotide ist). Komplexere Distanzmodelle wurden ebenfalls auf die Daten angewendet, mit ähnlichen Ergebnissen wie die, die durch p-Distanz präsentiert wurden (Daten nicht gezeigt). Die Distanzmatrix wurde verwendet, um ein Phylogramm mit der Nachbarverknüpfungsmethode (Saitou und Nei 1987) zu erstellen. Die Berechnungen der genetischen Distanzen und die Phylogramme wurden mit dem MEGA 3.0 Programm (Kumar et al. 2004) durchgeführt.
Wir haben auch Muster der intra-spezifischen Variation in D. antonietae durch Analysen der molekularen Varianz (AMOVAs; Excoffier et al. 1992) untersucht. Für die AMOVA wurden die Populationen von D. antonietae in 2 Gruppen eingeteilt: Atlantikküste und Paraná–Paraguay-Flussbecken. Die AMOVA wurde mit Arlequin 3.0 durchgeführt (Excoffier et al. 2005).
Ergebnisse und Diskussion
Sechsundzwanzig neue pBuM-2 Satelliten-DNA-Monomere wurden erhalten: 4 aus der Lokalität J56 (Zugangsnummer FJ935973– FJ93597G); 4 aus der Lokalität Via (Zugangsnummer FJt35t77–FJt35t80); 4 aus der Lokalität J26 (Zugangsnummer FJ935981–FJ935984); 3 aus der Lokalität J5/ (Zugangsnummer FJ935985–FJ935987); 5 aus der Lokalität J2/ (Zugangsnummer FJ935988–FJ935992); 2 aus der Lokalität J55 (Zugangsnummer FJ935993–FJ935994); und 4 aus der Lokalität J22 (Zugangsnummer FJ935995–FJ935998). Die hohe Nukleotidähnlichkeit zwischen den in dieser Arbeit erhaltenen Sequenzen und den zuvor beschriebenen (Kuhn und Sene 2005) bestätigt, dass sie zur Familie der pBuM-2 Satelliten-DNA gehören.
Alle erhaltenen Sequenzen weisen die für D. antonietae typische Primärstruktur auf und wurden im Phylogramm zusammen mit den zuvor beschriebenen pBuM-2-Sequenzen von D. antonietae (Abbildung 2) in denselben Zweig gruppiert, selbst die aus den Populationen innerhalb der Kontaktzone mit D. serido, wo eine Hybridisierung zwischen D. antonietae und D. serido mit Introgression von mitochondrialer DNA vorgeschlagen wurde (C.K.B Santos, F.M. Sene und M.H. Manfrin, unveröffentlichte Daten). Diese Situation könnte mit den Beobachtungen zusammenhängen, dass mitochondriale Gene tendenziell leichter introgressieren als die nukleären Sequenzen (Dorado et al. 1992; Arnold 1993). Die am häufigsten zitierten Erklärungen für diese Beobachtung stehen im Zusammenhang mit Prozessen der cytonukleären Ungleichgewichte (Arnold 1993) und der Sterilität von Hybridmännchen (Aubert und Solignac 1990).
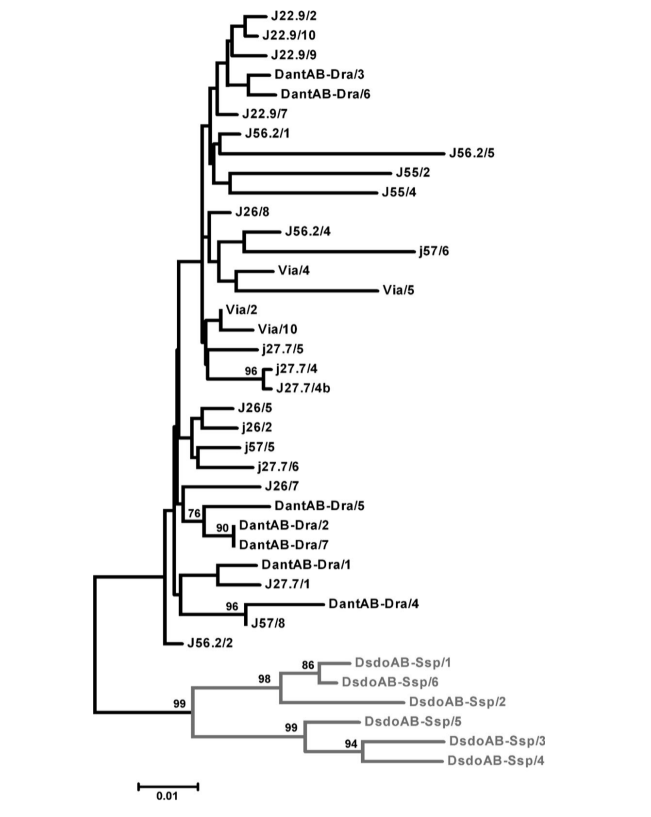
Es gibt ein hohes Maß an Sequenzähnlichkeit unter den pBuM-2-Sequenzen von D. antonietae (im Durchschnitt 95,7 %), und es wurden keine Mutationen gefunden, die für eine bestimmte Population diagnostisch wären. Die interpopulationale Variabilität war der intrapopulationalen Variabilität sehr ähnlich (Tabelle 1), und Sequenzen aus derselben Population gruppierten sich nicht in einem spezifischen Zweig im Dendrogramm (Abbildung 2). Die AMOVA, die mit den nach geografischen Gruppen (Paraná–Paraguay-Flussbecken und Atlantikküste) unterteilten Populationen durchgeführt wurde, zeigte, dass 99,30 % der gesamten genetischen Vielfalt der intragruppalen Variabilität zugeschrieben werden konnten, während nur 0,70 % der intergruppalen Variabilität zugeschrieben wurden (Tabelle 2). Der berechnete Фst war niedrig und hatte keine statistische Unterstützung (Tabelle 2).
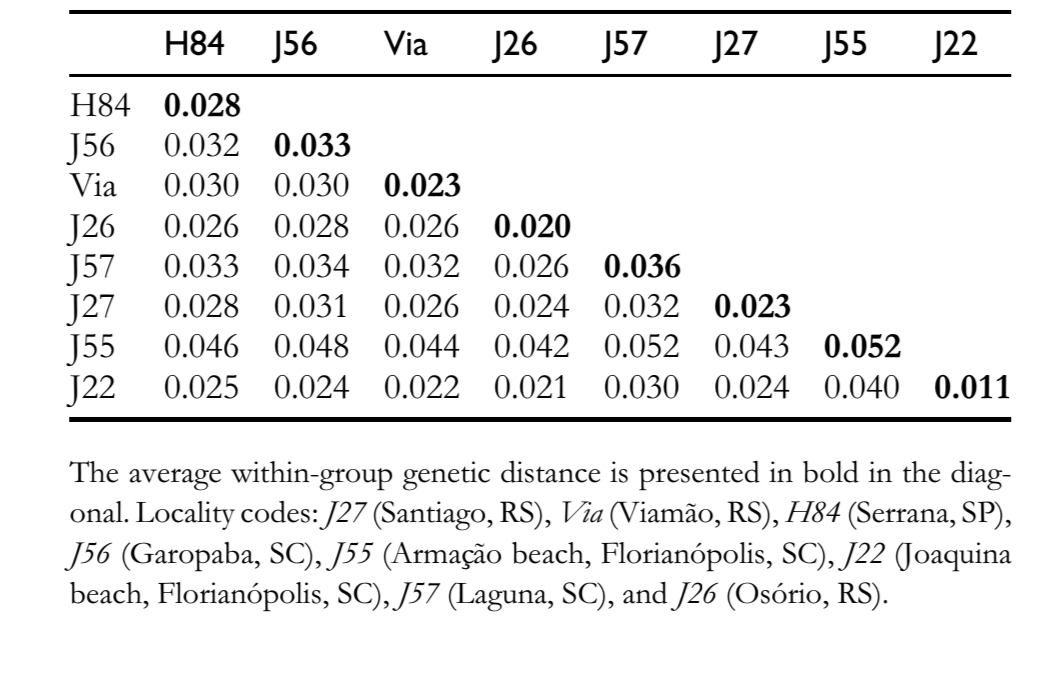

Die Populationen von D. antonietae aus dem Paraná–Paraguay-Flussbecken sind nach einem Modell der Isolation durch Distanz strukturiert (Monteiro und Sene 1995; Mateus und Sene 2007). Die beobachteten Chromosomeninversionen deuten auf eine Fragmentierung zwischen den Populationen von der Atlantikküste und denen aus dem Paraná–Paraguay-Flussbecken hin (Ruiz et al. 2000), während die mitochondrialen DNA-Daten nicht abschließend klären konnten, ob Fragmentierung oder Isolation durch Distanz die dominante Struktur war, die die Beziehung dieser 2 Populationen definierte (de Brito et al. 2002). Die hohe Nukleotidähnlichkeit von pBuM-2 in D. antonietae (Abbildung 2 und Tabellen 1 und 2) steht im Einklang mit der Aufrechterhaltung des Genflusses zwischen den Populationen dieser Art, selbst zwischen denen, die im Paraná–Paraguay-Flussbecken und an der Atlantikküste angesiedelt sind. Unsere Ergebnisse deuten darauf hin, dass die Isolation durch Distanz die mitochondriale DNA-Variabilität in D. antonietae erklären sollte; dieses Modell sagt einen Genfluss zwischen den Populationen voraus, wie aus den hier vorliegenden Satelliten-DNA-Daten abgeleitet. In diesem Kontext könnten die niedrigsten Chromosomeninversionspolymorphismen in den Populationen von der Atlantikküste von D. antonietae, die von Ruiz et al. (2000) festgestellt wurden, als Folge historischer und demografischer Ereignisse wie der Populationsausbreitung aus den Populationen der Täler des Paraná–Paraguay-Flussbeckens erklärt werden, wie durch mitochondriale DNA angedeutet (de Brito et al. 2002). Alternativ könnte diese Populationsdifferenzierung mit lokaler Anpassung an unterschiedliche Umgebungen in Anwesenheit von Genfluss verbunden sein, da Chromosomeninversionen möglicherweise mit adaptiven Merkmalen in kaktusliebenden Drosophila korreliert sind (Hasson et al. 1992; Fernández Iriarte et al. 2003). Eine alternative Erklärung für die hohe Nukleotidkonservierung von pBuM-2 in D. antonietae basiert auf den evolutionären Dynamiken von Satelliten-DNA-Sequenzen. Molekulare Mechanismen, die mit der Homogenisierung von tandemartig angeordneten Sequenzen zusammenhängen, wie ungleiche Crossing-over und Genkonversion durch einen Prozess, der als molekularer Antrieb bezeichnet wird (Dover 1982; Strachan et al. 1985), könnten auf die pBuM-Satelliten-DNA in dieser Art wirken. Unsere Analysen identifizierten keine Genkonversionsstrecken in den pBuM-2-Sequenzen, die aus D. antonietae isoliert wurden, was darauf hindeutet, dass dieser molekulare Mechanismus die Sequenzvielfalt von pBuM-2 in dieser Art nicht beeinflusst. Es wurde jedoch ein Minimum von 4 Rekombinationsevents in der Probe (Rm = 4) zwischen den Intervallen 130–136, 136–155, 174–376 und 376–378 festgestellt. Diese Rekombinationsevents könnten ein indirekter Beweis für ungleiche Crossing-over sein, das zur Aufrechterhaltung der Primärstruktur der pBuM-2-Sequenzen in den verschiedenen Populationen von D. antonietae beiträgt. Im Einklang mit dieser Hypothese enthält das D. antonietae-Genom eine große Anzahl von pBuM-2-Monomeren (Kuhn et al. 2007; Kuhn et al. 2008), was die Wahrscheinlichkeit ungleicher Crossing-over erhöht (Smith1976) und folglich Homogenisierung und konzertierte Evolution zur Folge hat.
Diese 2 alternativen Hypothesen, die die hohe Nukleotidähnlichkeit in den pBuM-2-Monomeren von D. antonietae erklären, schließen sich nicht gegenseitig aus. In diesem Zusammenhang schlagen wir vor, dass ein Gleichgewicht zwischen Genfluss zwischen Populationen und Homogenisierungsevents der tandemförmigen Satelliten-DNA-Arrays, gefördert durch ungleiche Crossing-over-Ereignisse, zur Erhaltung der primären Struktur von pBuM-2 in D. antonietae-Arten beiträgt.
Das Fehlen molekularer Divergenz in Satelliten-DNA-Sequenzen zwischen Populationen derselben Art, wie in der vorliegenden Arbeit festgestellt, wurde auch bei anderen Insektenarten beobachtet (Bachmann et al. 1994, 1998; Lorite et al. 2002; Feliciello et al. 2005). Zum Beispiel fanden Bachmann et al. (1994) hohe genetische Ähnlichkeiten unter den Monomeren des pDoP102-Satelliten in Populationen von Dolichopoda schiavazzii (Orthoptera), die laut allozymatischen Daten seit mindestens 235.000 Jahren voneinander isoliert sind. Laut diesen Autoren könnte die Homogenität in pDoP102 selbst in isolierten Populationen auf die Biologie der Satelliten-DNA zurückzuführen sein, da ungleiche Crossing-over und Genkonversion die Erhaltung der primären Struktur von Satelliten-DNA-Sequenzen induzieren können (Bachmann et al. 1994). In einer anderen Studie wurde das Vorhandensein von Genkonversionsstrecken in den Monomeren einer Satelliten-DNA-Familie als Beweis für Homogenisierungsevents in dieser Satelliten-DNA verwendet, was die hohe intraspezifische Erhaltung von Satelliten-DNA im phytophagen Käfer Xanthogaleruca luteola erklärt (Lorite et al. 2002).
In der D. buzzatii-Clusterart wurde auch eine hohe intraspezifische Erhaltung der pBuM-2-Sequenz in D. seriema (Kuhn und Sene 2004) und D. gouveai (Franco, Kuhn et al. 2006) identifiziert. Darüber hinaus sind die Populationen von D. buzzatii hinsichtlich der pBuM-1-Satelliten-DNA, einem weiteren Mitglied der pBuM-Familie (Kuhn et al. 2003), homogen. Bei D. seriema und D. buzzatii wurde angedeutet, dass das Fehlen von Populationsdifferenzierung in den Satelliten-DNA-Sequenzen auf vergangene und aktuelle Genflussereignisse zwischen den Populationen zurückzuführen ist, die mit den Ergebnissen übereinstimmen, die aus anderen genetischen Markern, die in denselben Populationen analysiert wurden, gewonnen wurden (Kuhn et al. 2003; Kuhn und Sene 2004). Für
D. gouveai, das nach einem „Insel“-Modell strukturiert ist (de Brito et al. 2002; Moraes und Sene 2007), war die Hauptannahme zur Erklärung der Erhaltung der primären Struktur in der pBuM-2 Satelliten-DNA, dass unabhängige Homogenisierungsmechanismen, die repetitive Einheiten, die unter den Populationen geteilt werden, begünstigen, nach dem Isolationsereignis der Populationen von D. gouveai (Franco, Kuhn et al. 2006) auftraten. Der D. buzzatii-Cluster besteht aus Schwesterarten, deren Taxonomie auf den morphometrischen Eigenschaften des Aedeagus basiert, der das intromittente Organ der männlichen Genitalien von Insekten ist (Vilela 1983; Tidon-Sklorz und Sene 2001; Franco, Prado et al. 2006). Somit erfolgt die Identifizierung von weiblichen Individuen des D. buzzatii-Clusters, die aus der natürlichen Umgebung stammen, durch die Identifizierung ihrer männlichen Nachkommen, was die Etablierung von Isofemale-Linien notwendig macht, was eine mühsame Aufgabe für cactophilische Drosophila-Arten sein kann. Darüber hinaus ist die korrekte Artendiskriminierung, die nur auf quantitativen Merkmalen wie der Morphologie des Aedeagus basiert, manchmal schwierig aufgrund der Anwesenheit morphologisch mehrdeutiger Individuen, insbesondere wenn die Individuen in Kontaktzonen zwischen Arten des D. buzzatii-Clusters gesammelt werden. In diesem Sinne hebt die starke intraspezifische Erhaltung der pBuM Satelliten-DNA-Sequenzen, die in mehreren Studien erhalten wurden (Kuhn et al. 2003; Kuhn und Sene 2004; Franco, Kuhn et al. 2006; vorliegendes Papier), selbst in genetisch strukturierten Arten, die Nützlichkeit von pBuM-2 zur Lösung taxonomischer Konflikte innerhalb der D. buzzatii-Clusterarten hervor, ein anerkanntes Modell für Studien der Evolutionsbiologie (Manfrin und Sene 2006).
Autoren: Fernando Faria Franco, Fabio Melo Sene, Maura Helena Manfrin
Referenzen:
- Arnold J. 1993. Cytonukleare Ungleichgewichte in Hybridzonen. Annu Rev Ecol Syst. 24:521–554.
- Aubert J, Solignac M. 1990. Experimentelle Beweise für die Mitochondrien-DNA-Introgression zwischen Drosophila-Arten. Evolution. 44:1272–1282.
- Bachmann L, Tomiuk J, Adis J, Vohland K. 1998. Genetische Differenzierung der Tausendfüßler Pycnotropis epiclysmus, die in saisonal überfluteten und nicht überfluteten Amazonaswäldern leben. J Zool Syst Evol Res. 36:65–70.
- Bachmann L, Venanzetti F, Sbordoni V. 1994. Charakterisierung einer artspezifischen Satelliten-DNA-Familie von Dolichopoda schiavazzi (Orthoptera, Rhaphidophoridae) Höhlenheuschrecken. J Mol Evol. 39:274–281.
- Betrán E, Rozas J, Navarro A, Barbadilla A. 1997. Die Schätzung der Anzahl und der Längendistibution von Genkonversionsstrecken aus DNA-Sequenzdaten der Population. Genetics. 146:89–99.
- Charlesworth B, Sniegowski P, Stephan W. 1tt4. Die evolutionären Dynamiken von repetitiver DNA in Eukaryoten. Nature. 371:215–220.
- de Brito AR, Manfrin MH, Sene FM. 2002. Verschachtelte kladistische Analyse brasilianischer Populationen von Drosophila serido. Mol Phylogenet Evol. 22: 131–143.
- Dorado O, Rieseberg LH, Arias D. 1992. Chloroplasten-DNA-Introgression in Sonnenblumen in Südkalifornien. Evolution. 46:566–572.
- Dover G. 1982. Molekularer Antrieb: ein kohäsiver Modus der Speziation. Nature. 299:111–117.
- Elder JF, Turner BJ. 1tt4. Konzertierte Evolution auf Populationsebene: Pupfish HindIII Satelliten-DNA-Sequenzen. Proc Natl Acad Sci U S A. 91: 994–998.
- Excoffier L, Laval G, Schneider S. 2005. Arlequin (Version 3.0): ein integriertes Softwarepaket zur Analyse von Populationsgenetikdaten. Evol Bioinform Online. 1:47–50.
- Excoffier L, Smouse PE, Quattro JM. 1992. Analysen der molekularen Varianz, die aus metrischen Abständen zwischen DNA-Haplotypen abgeleitet wurden: Anwendung auf Daten zur mitochondrialen DNA-Einschränkung des Menschen. Genetics. 131:479–491.
- Feliciello I, Picariello O, Chinali G. 2005. Die erste Charakterisierung der Gesamtvariabilität repetitiver Einheiten in einer Art zeigt unerwartete Merkmale von Satelliten-DNA. Gene. 349:153–164.
- Fernández Iriarte PJ, Norry FM, Hasson ER. 2003. Chromosomale Inversionen beeinflussen Körpergröße und -form in verschiedenen Fortpflanzungsressourcen von Drosophila buzzatii. Heredity. 91:51–59.
- Franco FF, Kuhn GCS, Sene FM, Manfrin MH. 2006. Erhaltung der pBuM–2 Satelliten-DNA-Sequenzen unter geografisch isolierten Drosophila gouveai Populationen aus Brasilien. Genetica. 128:287–295.
- Franco FF, Prado PRR, Sene FM, Costa LF, Manfrin MH. 200G. Morphologie des Aedeagus als diskriminierendes Merkmal in zwei eng verwandten kaktusliebenden Arten von Drosophila (Diptera; Drosophilidae) in Südamerika. An Acad Bras Cienc. 78:203–212.
- Franco FF, Sene FM, Manfrin MH. 2008. Molekulare Charakterisierung der SSS13t Satelliten-DNA-Familie in Geschwisterarten des Drosophila buzzatii Clusters. Genet Mol Biol. 31:155–159.
- Hall TA. 1999. BioEdit: ein benutzerfreundlicher biologischer Sequenzalignierungseditor und Analyseprogramm für Windows t5/t8/NT. Nucleic Acids Symp Ser. 41:95–98.
- Hasson E, Fanara JJ, Rodríguez C, Vilardi JC, Reig OA, Fontdevila A. 1992. Die evolutionäre Geschichte von Drosophila buzzatii. XXIV. Zweite Chromosomeninversionen haben unterschiedliche durchschnittliche Auswirkungen auf die Thoraxlänge. Heredity. 68:557–563.
- Hudson RR, Kaplan NL. 1985. Statistische Eigenschaften der Anzahl von Rekombinationsevents in der Geschichte einer Probe von DNA-Sequenzen. Genetics. 111:147–164.
- Kuhn GCS, Bollgönn S, Sperlich D, Bachmann L. 1999. Charakterisierung einer artspezifischen Satelliten-DNA von Drosophila buzzatii. J Zool Syst Evol Res. 37:109–112.
- Kuhn GCS, Franco FF, Manfrin MH, Moreira-Filho O, Sene FM. 2007. Niedrige Raten der Homogenisierung der DBC-150 Satelliten-DNA-Familie, die auf ein einzelnes Paar von Mikrochromosomen in Arten des Drosophila buzzatii Clusters beschränkt ist. Chromosome Res. 15:457–469.
- Kuhn GCS, Franco FF, Silva WA Jr, Martinez-Rossi NM, Sene FM. 2003. Zur Variabilität der pBuM18t Satelliten-DNA unter südamerikanischen Populationen von Drosophila buzzatii. Hereditas. 139:161–166.
- Kuhn GCS, Sene FM. 2004. Charakterisierung und interpopulationale Variabilität einer komplexen HpaI Satelliten-DNA von Drosophila seriema (repleta Gruppe). Genetica. 121:241–249.
- Kuhn GCS, Sene FM. 2005. Evolutionärer Wechsel von zwei pBuM Satelliten-DNA-Subfamilien im Drosophila buzzatii Artencluster (repleta Gruppe): von alpha zu alpha/beta Anordnungen. Gene. 349:77–85.
- Kuhn GCS, Sene FM, Moreira-Filho O, Schwarzacher T, Heslop-Harrison JS. 2008. Sequenzanalyse, chromosomale Verteilung und langfristige Organisation zeigen, dass der schnelle Wechsel neuer und alter pBuM Satelliten-DNA-Wiederholungen zu unterschiedlichen Mustern der Variation in sieben Arten des Drosophila buzzatii Clusters führt. Chromosome Res. 16:307–324.
- Kumar S, Tamura K, Nei M. 2004. MEGA3: integrierte Software für die Analyse molekularer evolutionärer Genetik und Sequenzalignment. Brief Bioinform. 5:150–163.
- Lorite P, Carrillo JA, Garneria I, Petitpierre E, Palomeque T. 2002. Satelliten-DNA im Ulmenblattkäfer, Xanthogaleruca luteola (Coleoptera, Chrysomelidae): Charakterisierung, interpopulationale Analyse und Chromosomenstandort. Cytogenet Genome Res. 98:302–307.
- Manfrin MH, Sene FM. 200G. Kaktusliebende Drosophila in Südamerika: ein Modell für evolutionäre Studien. Genetica. 126:57–75.
- Mateus RP, Sene FM. 2003. Temporale und räumliche Allozyme-Variation in der südamerikanischen kaktusliebenden Drosophila antonietae (Diptera; Drosophilidae). Biochem Genet. 41:219–233.
- Mateus RP, Sene FM. 2007. Populationsgenetische Studie der Allozyme-Variation in natürlichen Populationen von Drosophila antonietae (Insecta, Diptera). J Zool Syst Evol Res. 45:136–143.
- Monteiro SG, Sene FM. 1tt5. Morfometrische Studie von Populationen von Drosophila serido aus den zentralen und südlichen Regionen Brasiliens. Rev Bras Genet (Suppl). 18:283.
- Moraes EM, Sene FM. 2007. Mikrosatelliten- und morphometrische Variation in Drosophila gouveai: die relative Bedeutung historischer und aktueller Faktoren bei der Gestaltung der genetischen Populationsstruktur. J Zoolog Syst Evol Res. 45:336–344.
- Palomeque T, Lorite P. 2008. Satelliten-DNA bei Insekten: eine Übersicht. Heredity. 100:564–573.
- Pereira MAQR, Vilela CR, Sene FM. 1983. Hinweise zu Fortpflanzungs- und Futterplätzen einiger Arten der repleta-Gruppe der Gattung Drosophila (Diptera, Drosophilidae). Cienc Cult. 35:1313–1319.
- Picariello O, Feliciello I, Bellinero R, Chinali G. 2002. S1 Satelliten-DNA als taxonomisches Merkmal bei braunen Fröschen: molekulare Beweise, dass Rana graeca graeca und Rana graeca italica unterschiedliche Arten sind. Genome. 45: 63–70.
- Ruiz A, Cassian AM, Kuhn GCS, Alves MAR, Sene FM. 2000. Das Drosophila serido Speziationsrätsel: neue Teile zusammenfügen. Genetica. 108:217–227.
- Saitou N, Nei M. 1987. Die Nachbarverknüpfungsmethode: eine neue Methode zur Rekonstruktion phylogenetischer Bäume. Mol Biol Evol. 4:406–425.
- Sambrook J, Fritsh EF, Maniatis T. 1989. Molekulare Klonierung: ein Laborhandbuch. 2. Auflage. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press.
- Smith GP. 1976. Evolution wiederholter DNA-Sequenzen durch ungleiche Überkreuzung. Science. 191:528–535.
- Strachan T, Webb D, Dover GA. 1985. Übergangsphasen des molekularen Antriebs in mehrfachen DNA-Familien in Drosophila. EMBO J. 4:1701–1708.
- Thompson JD, Higgins DG, Gibson TJ. 1994. CLUSTAL W: Verbesserung der Sensitivität der progressiven multiplen Sequenzausrichtung durch Sequenzgewichtungen, positionsspezifische Gap-Strafen und Wahl der Gewichtsmatrix. Nucleic Acids Res. 22:4673–4680.
- Tidon-Sklorz R, Sene FM. 2001. Zwei neue Arten des Drosophila serido Geschwistersets (Diptera, Drosophilidae). Iheringia. 90:141–146.
- Ugarkovic D, Plohl M. 2002. Variation in Satelliten-DNA-Profilen – Ursachen und Auswirkungen. EMBO J. 21:5955–5959.
- Vilela CR. 1983. Eine Revision der Drosophila repleta Artengruppe (Diptera; Drosophilidae). Rev Bras Entomol. 27:1–114.
- Watabe H, Bachmann L, Haring E, Sperlich D. 1997. Taxonomische und molekulare Studien zu Drosophila sinobscura und D. hubeiensis, zwei Geschwisterarten der D. obscura Gruppe. J Zool Syst Evol Res. 35:81–94.
- Wu WL, Wang JP, Tseng MC, Chiang TY. 1999. Klonierung und genetische Variabilität einer HindIII repetitiven DNA in Acrossocheilus paradoxos (Cyprinidae). Genome. 42:780–788.
